Quero fazer um mapa de cluster / mapa de calor dos dados de presença-ausência de genes de pacientes nos quais os genes serão agrupados em categorias (por exemplo, quimiotaxia, endotoxina etc.) e rotulados adequadamente. Não encontrei nenhuma opção na documentação marítima. Eu sei como gerar o mapa de calor, apenas não sei como rotular os yticks como categorias. Aqui está uma amostra (não relacionada ao meu trabalho) do que eu quero alcançar:
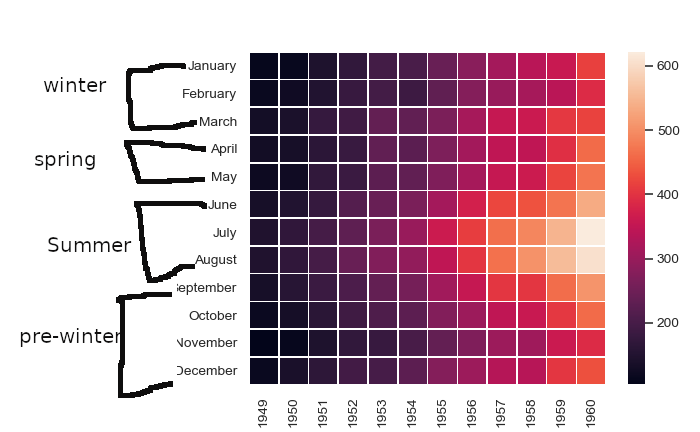
Aqui, yticklabels janeiro, fevereiro e março recebem o rótulo de grupo inverno e outros yticklabels também são rotulados de forma semelhante.

Respostas:
Reproduzi o exemplo que você deu no mar, adaptando a resposta de @ Stein daqui .
Dá:
Espero que ajude.
fonte
Ainda não testei isso com transoceânicos, mas o seguinte funciona com vanilla matplotlib.
fonte